【肝癌治疗领域新突破】运用单细胞测序技术揭示免疫反应与耐药机制的关键分子
2025-01-10
在肝癌治疗领域,我们正站在一个变革的十字路口。最新的研究突破(西奈山伊坎医学院的Josep M Llovet教授团队和鲁汶大学医院的Jeroen Dekervel教授团队共同完成)揭示了一种前沿的分子分型方法,它可能彻底改变我们对晚期肝细胞癌(HCC)患者治疗响应的理解。
关键词:晚期肝细胞癌;单细胞测序技术;肿瘤微环境;阿替利珠单抗与贝伐珠单抗组合治疗
1.研究的创新点
(1)首次将单细胞RNA测序的细胞特异性基因标签成功应用于批量RNA-seq数据,为解析治疗机制提供了新的方法。
(2)提出了“免疫活性”、“血管生成驱动”及“耐药”分子分类框架,为未来患者分层治疗提供了理论基础。
(3)阐明了NRP1低表达作为血管生成驱动亚型的潜在生物标志物,以及Notch和TGF-β在耐药中的重要作用。
2.背景知识
肝细胞癌(Hepatocellular Carcinoma, HCC)是全球癌症相关死亡的第三大原因,晚期HCC患者的系统性治疗选择较为有限。近年来,阿替利珠单抗(Atezolizumab)与贝伐珠单抗(Bevacizumab)的组合治疗(简称atezo+bev)通过IMbrave150 III期研究显示相较索拉非尼(Sorafenib)显著延长了生存期,成为晚期HCC的一线标准治疗方案。然而,针对atezo+bev疗效的分子机制及耐药原因尚未完全明确,尤其是在肿瘤微环境(TME)如何影响治疗结果方面。因此本研究旨在利用单细胞RNA测序(scRNA-seq)技术,识别晚期HCC患者中与atezo+bev治疗响应相关的分子特征,并探讨主要耐药机制。研究的最终目标是根据分子特征对患者进行分类,以指导个性化治疗策略。
3.研究设计
利用scRNA-seq高分辨率特点,生成适用于批量RNA测序(bulk RNA-seq)分析的基因特征,以此为基础重新分析大量样本的bulk RNA-seq数据结果,识别并研究晚期HCC中atezo+bev治疗的响应模式及耐药机制(图1)。
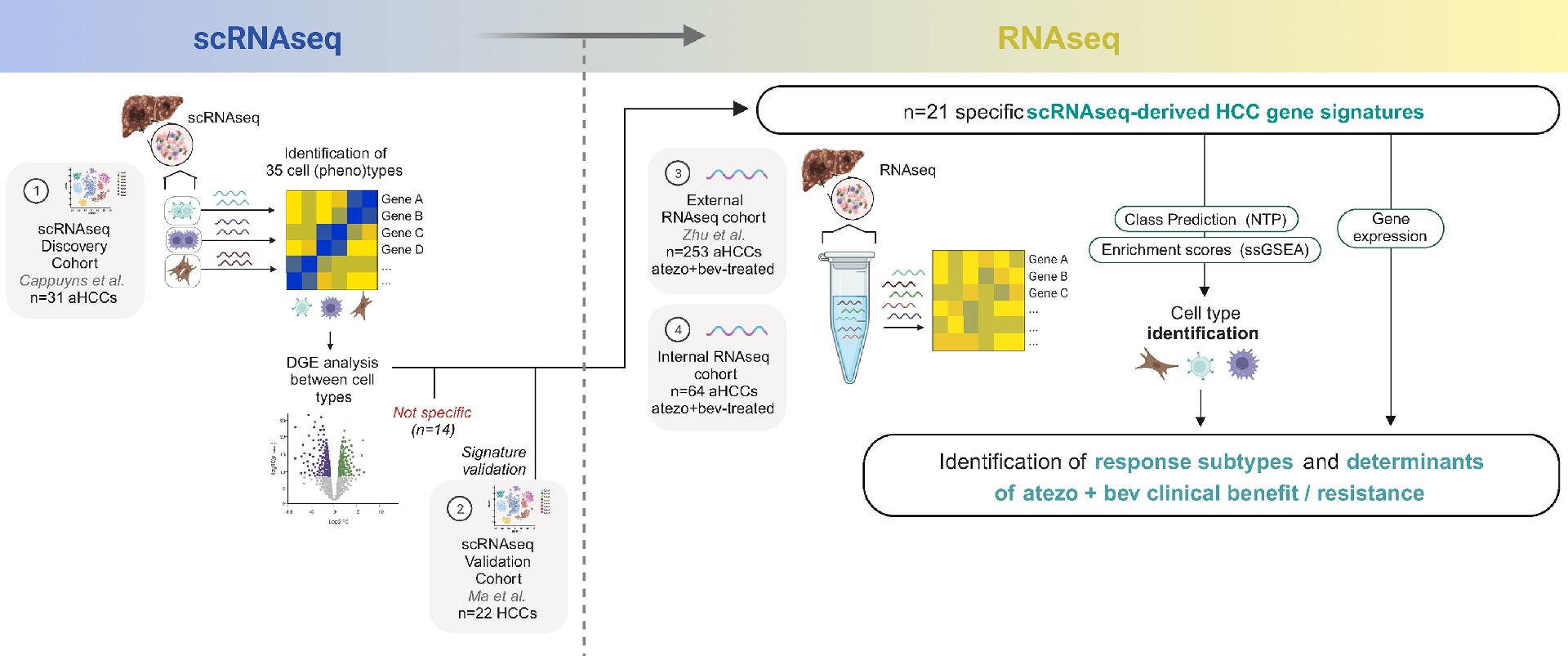
图1:研究方案设计
4.研究结果
(1)鉴定肿瘤组织细胞的基因特征
研究通过scRNA-seq数据,识别了晚期HCC肿瘤微环境中的35种细胞表型,包括恶性肝细胞、免疫细胞和间质细胞。每种细胞表型对应一组基因特征,这些基因特征通过差异表达分析获得,筛选了在特定细胞类型中高度表达的基因。这些基因的表达特征被验证为稳健且与相应细胞的功能一致。在验证阶段,使用另一个scRNA-seq队列,对所生成的基因特征进行验证,确保其在不同样本中的一致性。最终,研究确认了21种具有高特异性的基因特征,这些基因特征能够有效地代表晚期HCC中不同细胞表型的特征,具备了稳健性和生物学合理性,为进一步分析患者的治疗反应提供了可靠的分子工具(图2)。
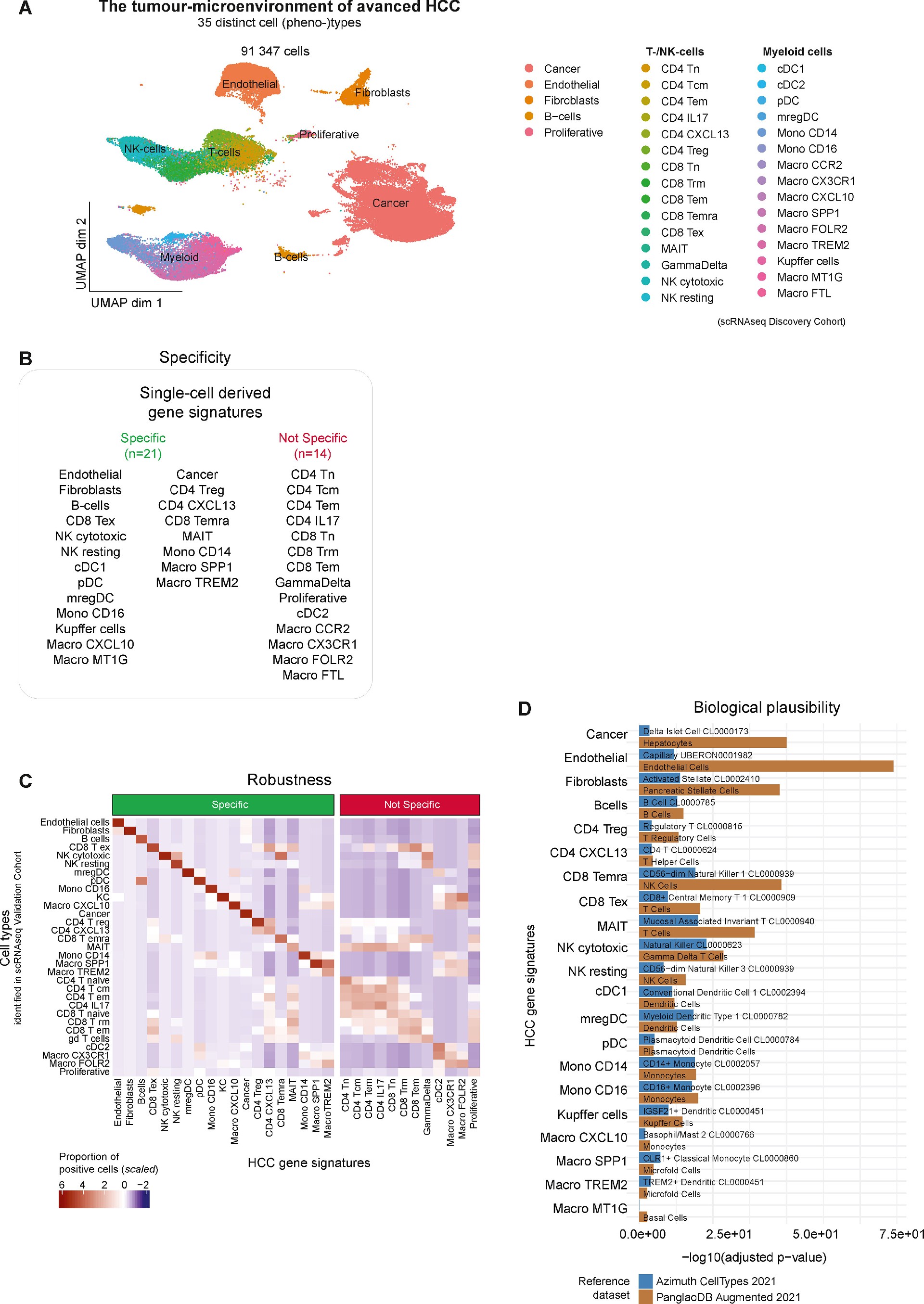
图2:鉴定HCC的单细胞基因特征
(2)HCC基因特征与晚期HCC中atezo+bev治疗反应的关系
研究利用从晚期HCC样本中生成的21种基因特征,分析了它们在422个HCC患者样本中的表达情况。这些患者接受了atezo+bev治疗,其中317名患者为主要治疗组,另外47名患者接受了阿替利珠单抗单药治疗,58名患者接受了索拉非尼治疗。通过分析基因特征的富集情况,研究揭示了免疫细胞特征在atezo+bev治疗中的重要性。
结果表明,CD8+效应T细胞(CD8 Temra和CD8 Tex)及CXCL10+巨噬细胞的富集是atezo+bev治疗反应的主要标志。这些细胞特征在atezo+bev响应者中普遍存在,且与较长的无进展生存期(PFS)相关。此外,这三种免疫细胞的富集预测了治疗的反应性,敏感性和特异性较高,尽管在一些患者中免疫反应较弱。研究还发现,免疫相关细胞群的富集与治疗效果密切相关,特别是CD8+效应T细胞和CXCL10+巨噬细胞的共存(图3)。通过这些基因标记,可以有效地识别atezo+bev的响应者,并为后续的临床治疗提供参考。
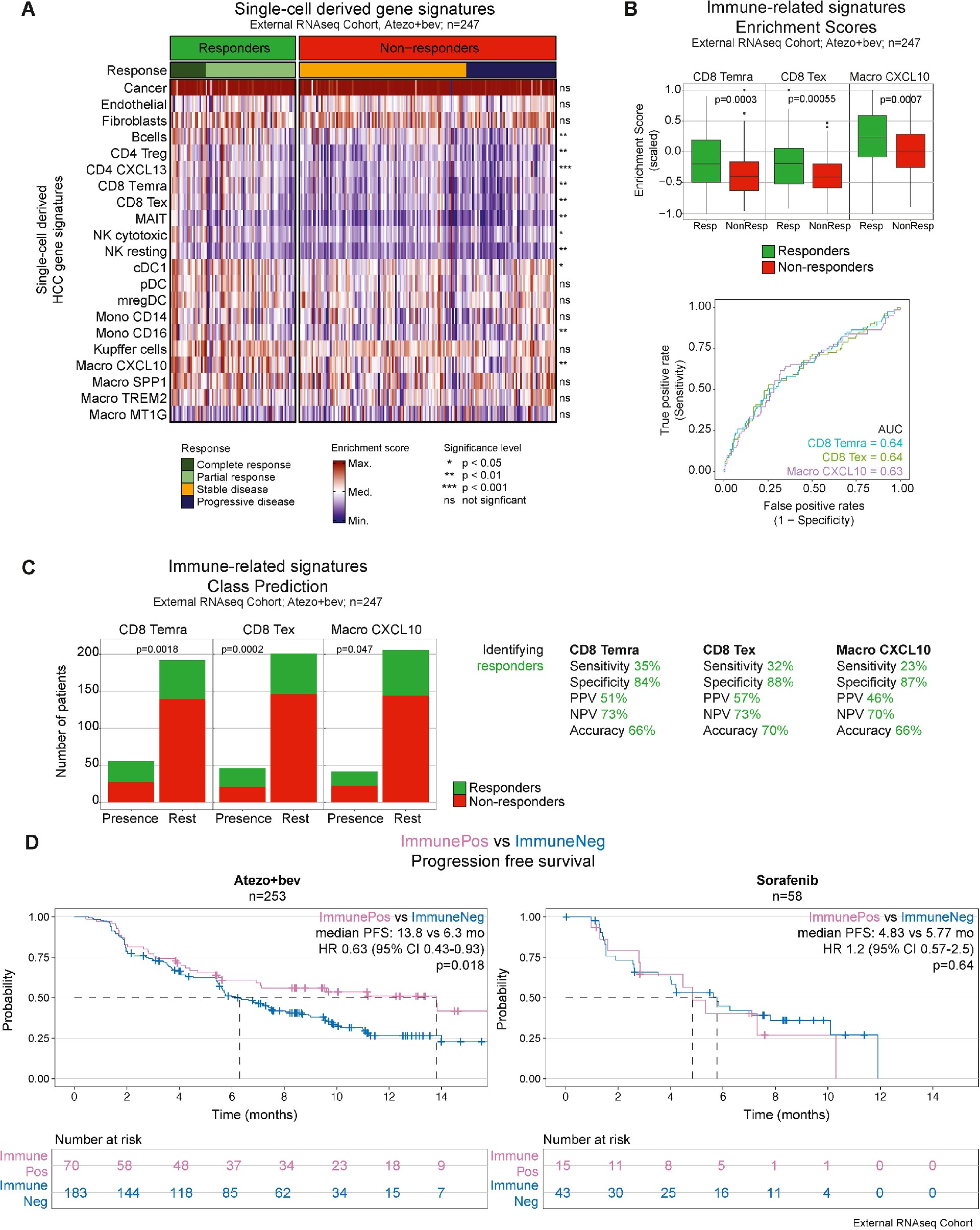
图3:HCC基因特征与atezo+bev治疗反应的关系
(3)免疫介导与非免疫介导的atezo+bev治疗反应在晚期HCC中的比较
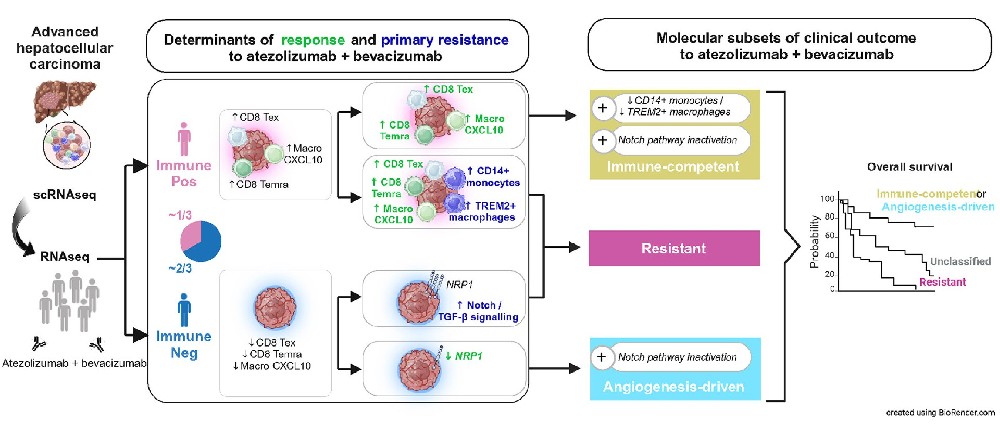
研究进一步分析了atezo+bev治疗的免疫介导反应与非免疫介导反应在晚期HCC中的区别。结果表明,约40%的atezo+bev响应者展示了强烈的免疫反应,这些肿瘤富含CD8+效应T细胞和CXCL10+巨噬细胞,呈现免疫活跃的肿瘤微环境。这些免疫相关的细胞群体与更长的无进展生存期相关,并且表现出典型的免疫检查点抑制剂(ICI)治疗反应特征(图4)。然而,剩余的60%的atezo+bev响应者缺乏上述免疫细胞的明显富集,反而表现出较强的基因组不稳定性,特别是TP53突变等特征,显示出抗血管生成的特性。与免疫富集的肿瘤不同,这些肿瘤在治疗反应中更多地依赖于抗血管生成作用,尤其是在低NRP1表达的情况下,这一特征与贝伐珠单抗的抗血管生成效应密切相关(图5)。此外,这部分患者的免疫细胞浸润程度较低,其肿瘤微环境呈现较低的免疫反应,但表现出较高的基因组不稳定性和多种与抗血管生成相关的特征。该研究揭示了免疫介导与非免疫介导的治疗反应机制之间的差异,为未来的治疗策略提供了重要的参考。

图4:晚期HCC患者对atezo+bev的免疫介导反应
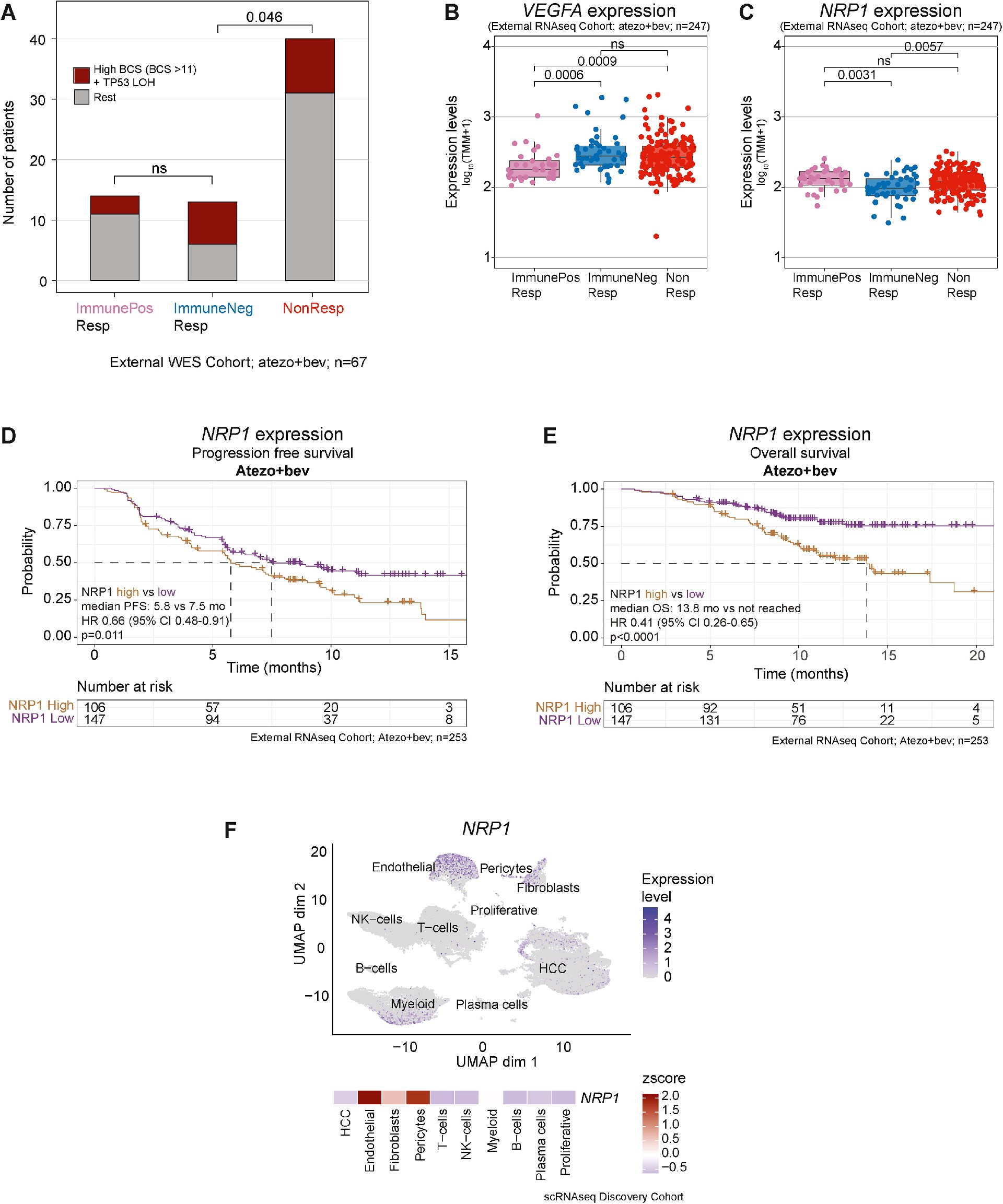
图5:晚期HCC患者对atezo+bev的血管生成相关反应
(4)免疫抑制性髓系细胞和间质细胞群体造成atezo+bev耐药
研究发现,免疫抑制性髓系细胞和间质细胞群体在atezo+bev耐药中发挥了关键作用。具体而言,CD14+单核细胞和TREM2+巨噬细胞是耐药性的重要特征,它们富集在治疗无效的患者肿瘤中。这些免疫抑制性细胞通过抑制T细胞的免疫反应,可能削弱了atezo+bev的治疗效果。此外,研究还揭示了Notch和TGF-β信号通路的激活与耐药性密切相关。特别是,在耐药患者的肿瘤微环境中,这些信号通路的激活促进了免疫抑制性细胞的招募和功能,从而阻碍了免疫治疗的效果。通过机器学习方法,研究进一步验证了免疫抑制性细胞特征与耐药性之间的关联,为耐药机制的识别提供了新的分子标志物(图6)。
该发现为克服atezo+bev的耐药性提供了潜在的靶向策略,未来可能通过干预这些免疫抑制性细胞和关键信号通路,增强治疗的效果并改善患者的预后。

图6:晚期HCC患者对atezo+bev产生原发性耐药性的决定因素
(5)决定晚期HCC患者临床疗效的分子亚型
研究通过综合分析反应与耐药机制,提出了三种分子亚型,这些亚型能够预测atezo+bev治疗在晚期HCC中的临床获益。这些亚型分别是“免疫活性型”(Immune-competent)、“血管生成驱动型”(Angiogenesis-driven)和“耐药型”(Resistant)。免疫活性型和血管生成驱动型患者在接受atezo+bev治疗时表现出显著的生存获益,且总体生存期(OS)和无进展生存期(PFS)均明显延长。这些分子亚型的划分是基于肿瘤微环境中免疫细胞、免疫抑制性细胞、Notch信号通路活化以及NRP1表达水平等多个特征。免疫活性型患者肿瘤微环境免疫活跃,富含CD8+效应T细胞和CXCL10+巨噬细胞,而血管生成驱动型则表现出低NRP1表达,提示其主要受贝伐珠单抗的抗血管生成作用影响。耐药型患者则显示出免疫细胞缺乏或免疫抑制性细胞富集的特征,并伴随Notch和TGF-β信号通路的激活。该研究为根据分子特征进行患者分层提供了有力支持,并为未来的个性化治疗方案制定提供了理论基础。通过这些分子亚型的划分,能够更加准确地预测哪些患者能从atezo+bev治疗中受益,哪些则可能面临耐药挑战。
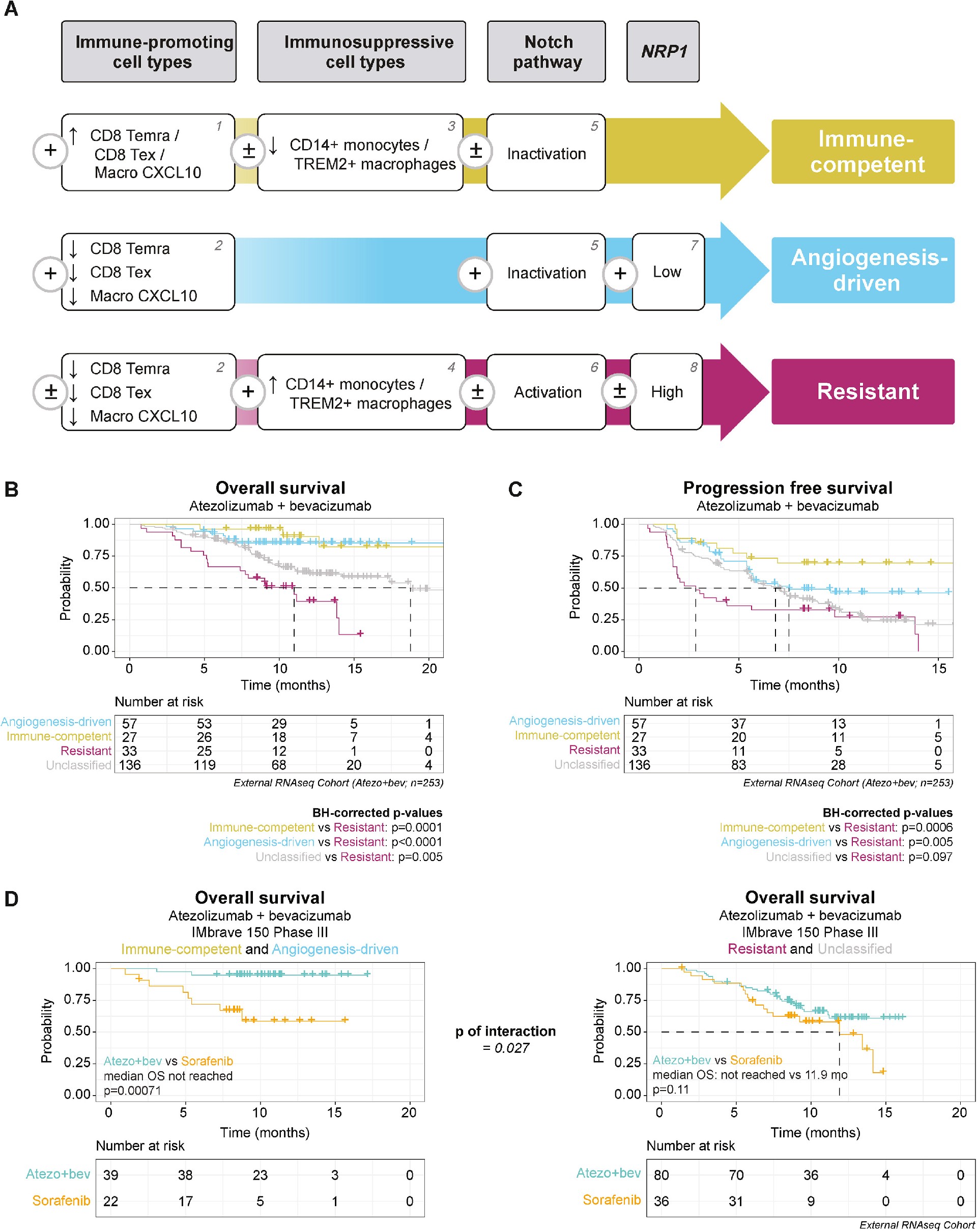
图7:分子亚群决定atezo+bev对晚期HCC的临床疗效
5.论文给我们的启示
(1)精准医疗的实践方向:本研究强调了基于分子机制的精准分层治疗在提高疗效方面的潜力。
(2)未来研究方向:进一步开发基于NRP1的生物标志物,用于预测患者对atezo+bev的响应;探索联合靶向免疫抑制性信号通路(如Notch和TGF-β)的新策略以克服耐药。
(3)临床应用的挑战:尽管研究提供了详细的分子机制,但部分生物标志物的临床验证仍需更大规模的前瞻性研究。
参考文献
1. Cappuyns S, Piqué-Gili M, Esteban-Fabró R, et al. Single-cell RNA seq-derived signatures define response patterns to atezolizumab + bevacizumab in advanced hepatocellular carcinoma. J Hepatol. (DOI:10.1016/j.jhep.2024.12.016)
2. Rimassa L, Finn RS, Sangro B. Combination immunotherapy for hepatocellular carcinoma. J Hepatol. 2023;79(2):506-515. (DOI:10.1016/j.jhep.2023.03.003)
3. Greten TF, Villanueva A, Korangy F, et al. Biomarkers for immunotherapy of hepatocellular carcinoma. Nat Rev Clin Oncol. 2023;20(11):780-798. (DOI:10.1038/s41571-023-00816-4)
4. Hwangbo H, Patterson SC, Dai A, Plana D, Palmer AC. Additivity predicts the efficacy of most approved combination therapies for advanced cancer. Nat Cancer. 2023;4(12):1693-1704. (DOI:10.1038/s43018-023-00667-z)
 技术咨询:
技术咨询: